
octobre 8, 2020 - Théo Marcille
Chez Sêmeia, on a la chance de pouvoir travailler sur l’une des bases de données médicales la plus complète au monde : les données de la Sécurité Sociale. En plus d’être exhaustive, cette base est structurée, documentée, et contient moins d’erreurs que la très (très) grande majorité des jeux de données qui peuvent être rencontrées en entreprise par des data scientist.
Concrètement, après une demande faite à la CNAM (Caisse nationale de l'assurance maladie), on reçoit sous forme de fichiers CSV des données extraites des bases du SNDS (Système National des Données de Santé). Données qui concernent un nombre d’individus variables en lien avec une pathologie spécifique, pour une plage de temps variable, et qui sont bien sûr pseudonymisés afin de rendre plus difficile l'identification des individus. Ces fichiers sont ensuite stockés sur un environnement sécurisé auquel se connectent les data scientist pour travailler.
Point positif : le jeu de données reçu sous forme de fichier CSV est très complet. Point négatif : le jeu de données reçu sous forme de fichier CSV est *très* complet. On parle en effet de plusieurs dizaines de fichiers par année, avec des relations entre eux parfois complexe, ce qui implique de devoir intégrer à la librairie Sêmeia un module de traitement initial des données.

Ce module a pour rôle de retraiter les données contenues dans les fichiers CSV et nécessaires au travail des data scientist dans un format plus pratique pour eux : découpées par *batch* de n individus pour éviter les problèmes de manque de RAM, et en ayant ajouté via des jointures toutes les informations nécessaires pour pouvoir effectuer la suite de l’étape de *feature engineering*. Exemple simple : le module va ajouter au fichier contenant les informations sur les diagnostics d’un séjour à l’hôpital les dates de ce séjour, qui se trouvaient dans un autre fichier.
Ce module, appelé en interne le **Data Handler** (DH pour les intimes), était à l’origine écrit uniquement en python, à l’aide de la librairie Pandas. Mais lorsque les projets ont commencé à être plus ambitieux, et qu’un seul fichier CSV peut dépasser la centaine de gigas, ce n’était plus vraiment suffisant. Pandas n’est, en effet, pas conçu pour charger des fichiers qui ne tiennent pas en RAM, et encore moins pour faire une jointure entre deux fichiers qui ne tiennent pas en RAM. Une réécriture complète du Data Handler a donc été entreprise pour permettre le passage à l’échelle.
En faisant un tour d’horizon des options qui s’offraient à nous, il y en a deux qui se sont détachées :
- Dask, une librairie python qui est principalement utilisée pour la parallélisation de calculs mais qui permet aussi de faire des traitements par lots de fichiers (plus de problèmes de RAM si le fichier n’est jamais chargé entièrement en mémoire)
- La solution « à l’ancienne », c’est-à-dire insérer les données dans une base de données relationnelle (avec en l’occurrence PostgreSQL comme SGBD), qui gère sans problème les opérations de jointure sur des jeux de données de cette taille.
En réalité, Dask ne fonctionnait pas correctement sur l’environnement sécurisé sur lequel sont stockées les données. PostgreSQL déclaré vainqueur par abandon.
Histoire d’avoir quelques garanties avant de se lancer dans la réécriture du Data Handler, tout un ensemble de benchmarks et de tests ont été réalisé avec des données synthétiques afin de vérifier que l’ensemble des fonctionnalités de l’ancien module étaient reproductibles avec une base de données, mais aussi que nous n’aurions pas de mauvaise surprise en ce qui concerne les temps de traitement.
Par exemple, on peut comparer le temps nécessaire pour rechercher les informations sur un ensemble précis de patients dans la base :
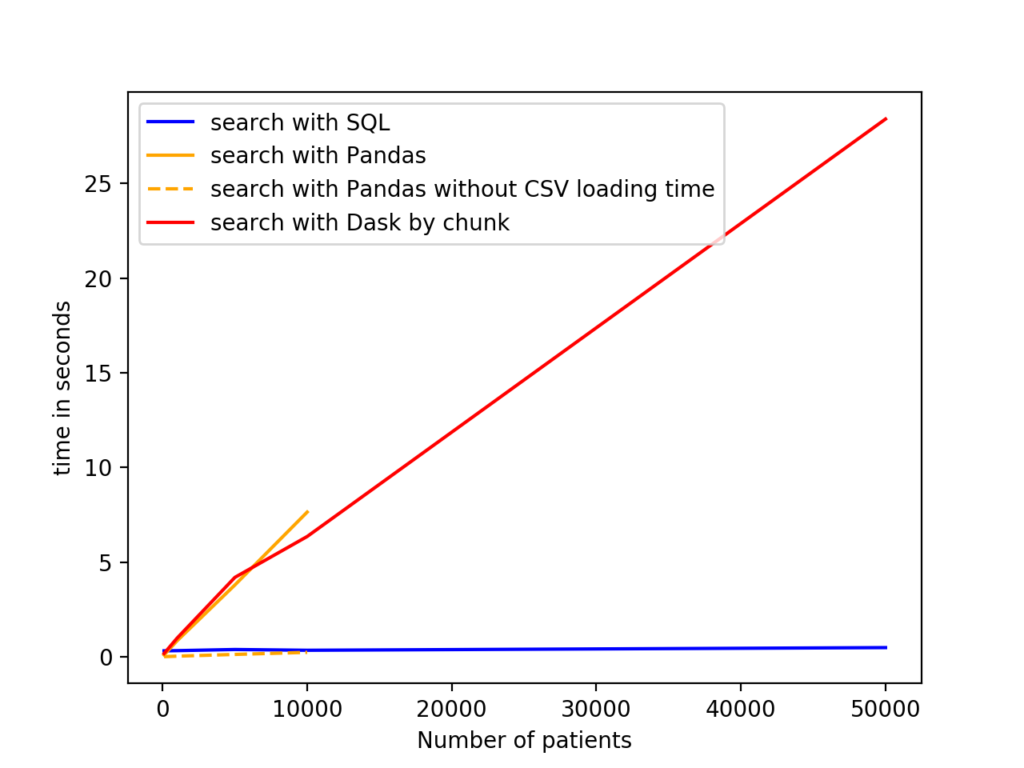
Le résultat est relativement clair : Pandas n’est pas suffisamment scalable pour être une alternative viable, tandis que Dask (s'il était utilisable) ne peut simplement
pas rivaliser avec la base PostgreSQL et son indexation des données.
Un petit détail fait cependant que la comparaison n’était pas totalement équitable : le graphe ne prend pas en compte le temps nécessaire à l’insertion des données
dans la base PostgreSQL, tandis que le temps de chargement des CSV est compté pour Pandas et Dask. Mais puisque cette insertion n’est réalisé qu’une seule fois
par jeu de données, PostgreSQL reste a terme largement plus efficace. Pour manipuler la BDD depuis la librairie, on utilise SQLAlchemy. Déjà parce qu’elle permet
une grande flexibilité des requêtes (juste ce que l’on recherche pour que l’utilisateur puisse extraire exactement les informations qu’il veut de la base de données),
mais aussi parce que ça permet d'éviter aux data scientist de devoir passer sans arrêt du Python au SQL. Le but final est simple : récupérer une table pré-traitée = un
seul appel de fonction, sans avoir à trop se soucier de gérer la connexion à la BDD ou de la manière dont sont stockées les données sur celle-ci.
Maintenant que les éléments techniques qui composerons le nouveau Data Handler ont été validés, comment peut on l’implémenter en pratique ?
Première étape : créer les tables. Comme on l’a vu précédemment, chaque jeu de données est composé de plusieurs dizaines de tables, qui comportent chacune un nombre non-négligeable de colonnes. Autant dire que créer manuellement toutes ces tables n’était pas une idée très réjouissante. Mais heureusement, les très sympathiques personnes du Health Data Hub ont eu la bonne idée de mettre à disposition sur leur [Gitlab](https://gitlab.com/healthdatahub) une description de toutes les tables du SDNS dans un format qui suit la [spécification Table Schema](https://specs.frictionlessdata.io/table-schema/). Et puisqu’une bonne nouvelle n’arrive jamais seule, il existe même une librairie Python appelée tableschema-sql qui peut créer automatiquement les tables de la base de données à partir de ces schémas descriptifs. Boum, notre base est prête à recevoir les données. Précision importante : l’automatisation était d’autant plus nécessaire que chaque jeu de données doit être stocké sur un serveur différent puisque les croisements entre les différents jeux de données sont interdits. Une nouvelle BDD doit donc être créée pour chaque projet.
Deuxième étape : remplir les tables. Avec quelques lignes de code et un usage intensif de la commande COPY FROM de PostgreSQL (qui est au passage beaucoup rapide que d’insérer les données ligne par ligne), on obtient une insertion automatique des données dans les tables correspondantes.
On ajoute à ça quelques fonctions pour simplifier la création d’index et l’optimisation de la base de données selon le projet pour lequel elle va être utilisée, une gestion des données réparties sur plusieurs années et des quelques spécificités des données médicales du SNDS, et voilà. Les data scientist peuvent transformer les fichiers CSV en une base de données prête a être utilisée avec seulement quelques appels aux fonctions du Data Handler.
Maintenant que nous avons notre BDD contenant toutes les données de la cohorte, il est l’heure d’utiliser sérieusement les possibilités offertes par cette base.
L’idée est de faciliter le travail des data scientist en appliquant tout un tas d’opérations aux données avant de la leur fournir. En effet, on va produire en sortie des fichiers contenant les mêmes informations que les tables, mais avec quelques modifications pour les rendre plus faciles à manipuler. Le nombre d’individus est trop important ce qui cause des problèmes de mémoire vive pendant l’étape de feature engineering ? On découpe les données en batch de n patients. Les tables ont besoin pour être utilisables d’informations situées dans d’autres tables ? On va effectuer les jointures nécessaires pour avoir en sortie des tables utilisables en *standalone*. Et puisque certaines tables ont besoin d’opération de filtrage spécifiques, le module va aussi s’en occuper automatiquement.
Exemple concret : le data scientist a besoin des informations sur les séjours à l'hôpital des individus, informations qui se trouvent dans une des tables de la base de données. Le module va d’abord appliquer un filtre sur la table pour exclure certains séjours précis (filtre standardisé par la Cnam). Mais, en l’état, la table est difficilement utilisable puisqu’elle ne comporte aucune information sur les individus en eux-mêmes (âge, sexe, etc.) qui sont elles situées dans une autre table. Le Data Handler va donc effectuer les jointures nécessaires entre ces deux tables (voir avec une troisième table si nécessaire) pour reconstituer des informations utilisables par le Data Scientist. Le tout de manière transparente pour l’utilisateur qui a juste à indiquer qu’il veut les informations sur les séjours à l’hôpital.

C'est là que le fait d'utiliser une librairie comme SQLAlchemy devient très pratique. En effet, les requêtes SQL sont représentées par des objets auxquels on peu appliquer des méthodes renvoyant elles-mêmes une requête. Par exemple, si `query` est un objet représentant une requête, `query.order_by(Table.column)` renvoie la requête à laquelle on a ajouté un `ORDER BY`. Le Data Handler essaye de tirer parti au maximum de cela, en construisant des fonctions qui servent de briques de bases et qui sont utilisées ou non (selon les informations demandées par le data scientist) pour construire la requête finale.
Une fois en production, la refonte du Data Handler a donc permis aux data scientist de Sêmeia de faire des études sur des cohortes beaucoup plus importantes que précédemment, et ce sans sacrifier la flexibilité de la librairie.